Eine neue Waffe für die Tuberkulosediagnostik und den Nachweis von Arzneimittelresistenzen: Eine neue Generation zielgerichteter Sequenzierung (tNGS) kombiniert mit maschinellem Lernen zur Diagnose von Tuberkulose-Überempfindlichkeit
Literaturbericht: CCa: ein auf tNGS und maschinellem Lernen basierendes Diagnosemodell, das sich für Menschen mit weniger bakterieller Tuberkulose und tuberkulöser Meningitis eignet.
Titel der Dissertation: Tuberkulose-gerichtete Next-Generation-Sequenzierung und maschinelles Lernen: eine ultrasensitive Diagnosestrategie für paucific pulmonale Tubuli und tubuläre Meningitis.
Zeitschrift: „Clinica Chimica Acta“
IF: 6,5
Veröffentlichungsdatum: Januar 2024

In Zusammenarbeit mit der Universität der Chinesischen Akademie der Wissenschaften und dem Beijing Chest Hospital der Capital Medical University entwickelte Macro & Micro-Test ein Tuberkulose-Diagnosemodell. Dieses basiert auf der neuen Generation der zielgerichteten Sequenzierung (tNGS) und maschinellem Lernen und ermöglicht eine extrem hohe Nachweisempfindlichkeit für Tuberkulose mit geringer Bakterienlast sowie für tuberkulöse Meningitis. Es bietet eine neue, hochsensitive Diagnosemethode für die klinische Diagnostik dieser beiden Tuberkuloseformen und trägt zu einer präzisen Diagnose, dem Nachweis von Arzneimittelresistenzen und der Behandlung von Tuberkulose bei. Gleichzeitig wurde festgestellt, dass zellfreie DNA (cfDNA) aus dem Patientenplasma als geeignetes Probenmaterial für die klinische Probenentnahme bei der Diagnose von tuberkulöser Meningitis (TBM) verwendet werden kann.
In dieser Studie wurden 227 Plasma- und Liquorproben verwendet, um zwei klinische Kohorten zu erstellen. Die Proben der labordiagnostischen Kohorte dienten der Entwicklung eines maschinellen Lernmodells zur Tuberkulose-Diagnostik, während die Proben der klinischen Kohorte zur Validierung des entwickelten Modells verwendet wurden. Alle Proben wurden zunächst mit einem speziell entwickelten Pool zielgerichteter Capture-Sonden für Mycobacterium tuberculosis angefärbt. Anschließend wurde basierend auf den TB-tNGS-Sequenzierungsdaten ein Entscheidungsbaummodell verwendet, um eine 5-fache Kreuzvalidierung der Trainings- und Validierungsdatensätze der labordiagnostischen Warteschlange durchzuführen und die diagnostischen Schwellenwerte für Plasma- und Liquorproben zu ermitteln. Die ermittelten Schwellenwerte wurden in zwei Testdatensätzen der klinischen Diagnose-Warteschlange zur Detektion eingesetzt, und die diagnostische Leistungsfähigkeit des Lernmodells wurde mittels ROC-Kurve bewertet. Abschließend wurde das Diagnosemodell für Tuberkulose erstellt.
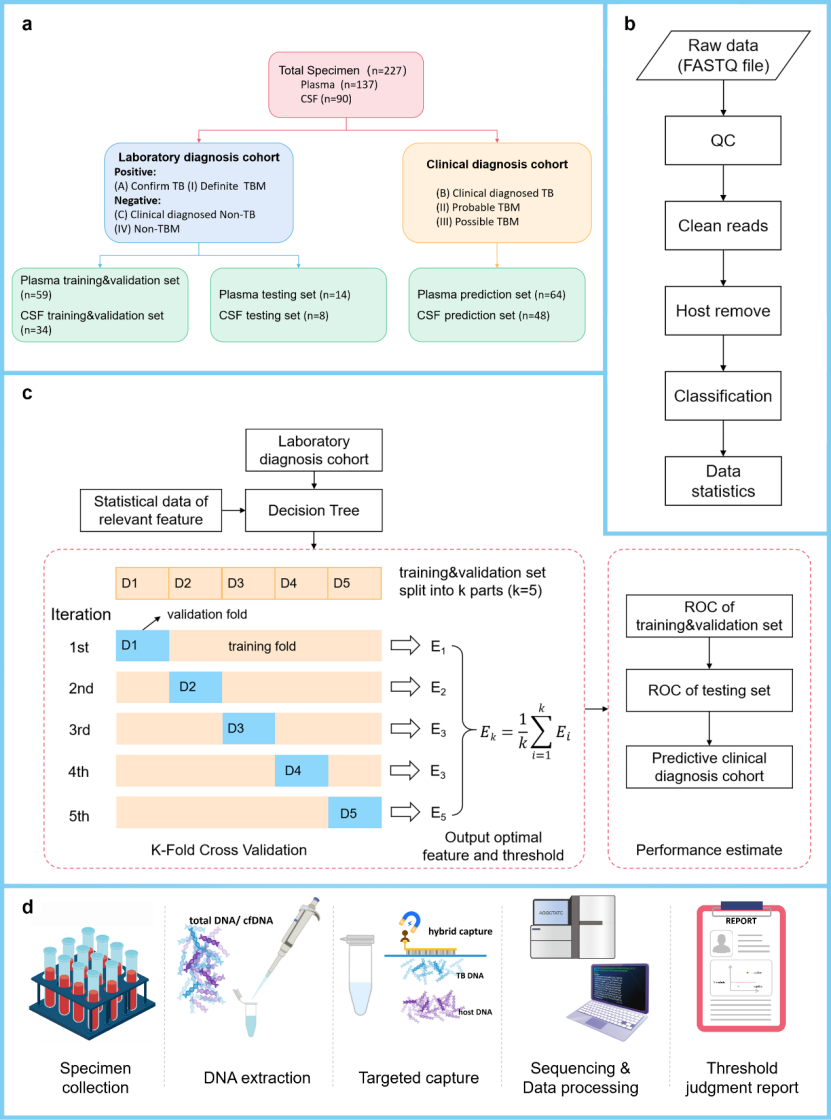
Abb. 1 schematische Darstellung des Forschungsdesigns
Ergebnisse: Gemäß den in dieser Studie ermittelten spezifischen Schwellenwerten für CSF-DNA (AUC = 0,974) und Plasma-cfDNA (AUC = 0,908) betrug die Sensitivität der CSF-Probe bei 227 Proben 97,01 % und die Spezifität 95,65 %. Für die Plasma-Probe lagen die entsprechenden Werte bei 82,61 % bzw. 86,36 %. Die Analyse von 44 gepaarten Plasma-cfDNA- und Liquorproben von Patienten mit tuberkulöser Meningitis (TBM) ergab eine hohe Übereinstimmung von 90,91 % (40/44) und eine Sensitivität von 95,45 % (42/44). Bei Kindern mit Lungentuberkulose ist die diagnostische Strategie dieser Studie bei Plasmaproben sensitiver als die Xpert-Nachweisergebnisse von Magensaftproben derselben Patienten (28,57 % vs. 15,38 %).
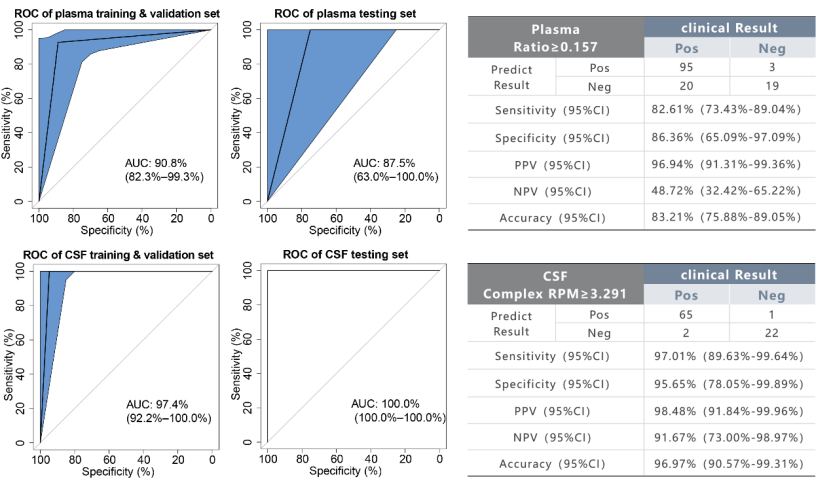
Abb. 2 Analyse der Leistungsfähigkeit des Tuberkulose-Diagnosemodells für Bevölkerungsproben
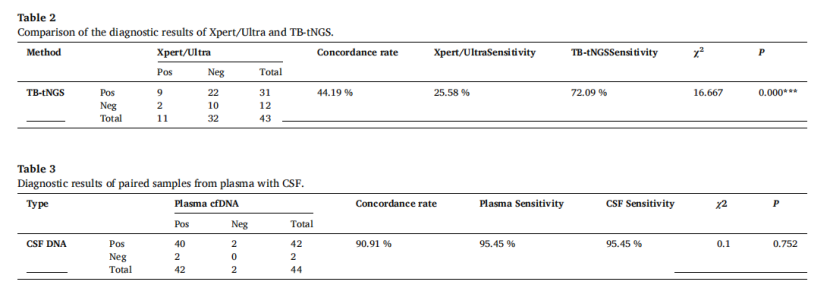
Abb. 3 Diagnostische Ergebnisse von gepaarten Proben
Schlussfolgerung: In dieser Studie wurde eine hochsensitive Diagnosemethode für Tuberkulose etabliert, die ein Diagnoseinstrument mit höchster Nachweisempfindlichkeit für klinische Patienten mit oligobakterieller Tuberkulose (negative Kultur) darstellt. Der Nachweis von hochsensitiver Tuberkulose mittels zellfreier DNA (cfDNA) im Plasma könnte eine geeignete Probenart für die Diagnose aktiver Tuberkulose und tuberkulöser Meningitis sein (Plasmaproben sind bei Verdacht auf Hirntuberkulose leichter zu gewinnen als Liquor).
Original-Link: https://www.sciencedirect.com/science/article/pii/s0009898123004990? via%3Dihub
Kurze Einführung in die Tuberkulose-Nachweisprodukte der Macro & Micro-Test-Serie
Angesichts der komplexen Probensituation von Tuberkulosepatienten und der vielfältigen Anforderungen bietet Macro & Micro-Test ein umfassendes Portfolio an NGS-Lösungen für die Extraktion von Sputumproben mittels Flüssigchromatographie, die Qualcomm-Bibliothekspräparation und Sequenzierung sowie die Datenanalyse. Die Produkte umfassen die Schnelldiagnostik von Tuberkulosepatienten, den Nachweis von Arzneimittelresistenzen bei Tuberkulose, die Typisierung von Mycobacterium tuberculosis und NTM, die Hypersensitivitätsdiagnostik bei bakteriennegativer Tuberkulose und bei Tuberkulosepatienten usw.
Serientests zum Nachweis von Tuberkulose und Mykobakterien:
| Artikelnummer | Produktname | Produkttestinhalte | Probenart | anwendbares Modell |
| HWTS-3012 | Probenfreigabemittel | Wird bei der Verflüssigungsbehandlung von Sputumproben eingesetzt und hat die erstklassige Registrierungsnummer Sutong Machinery Equipment 20230047 erhalten. | Sputum | |
| HWTS-NGS-P00021 | Qualcomm-Kit zur Mengenbestimmung bei hypersensitiver Tuberkulose (Sondenerfassungsmethode) | Nichtinvasiver (flüssigbioptischer) Nachweis von Überempfindlichkeitsreaktionen bei bakteriennegativer Lungentuberkulose und Hirnknoten; Die Proben von Personen, bei denen der Verdacht auf eine Infektion mit Tuberkulose oder nichttuberkulösen Mykobakterien bestand, wurden mittels Metagenomik mit hoher Sequenziertiefe analysiert. Dabei wurden Informationen zum Nachweis einer Infektion mit Tuberkulose oder nichttuberkulösen Mykobakterien sowie zu den wichtigsten Resistenzen von Mycobacterium tuberculosis gegen Erstlinienmedikamente bereitgestellt. | Peripheres Blut, alveoläre Lavageflüssigkeit, Hydrothorax und Aszites, Punktionsprobe, Liquor cerebrospinalis. | Zweite Generation |
| HWTS-NGS-T001 | Kit zur Typisierung von Mykobakterien und zum Nachweis von Arzneimittelresistenzen (Multiplex-Amplifikationssequenzierungsverfahren) | Mykobakterien-Typisierungstest, einschließlich MTBC und 187 NTM;Die Erkennung von Arzneimittelresistenzen bei Mycobacterium tuberculosis umfasst 13 Medikamente und 16 Kernmutationsstellen von Arzneimittelresistenzgenen. | Sputum, alveoläre Lavageflüssigkeit, Hydrothorax und Aszites, Punktionsprobe, Liquor cerebrospinalis. | Dualplattform der zweiten/dritten Generation |
Highlights: HWTS-NGS-T001 Mykobakterien-Typisierungs- und Resistenznachweiskit (Multiplex-Amplifikationsmethode)
Produkteinführung
Das Produkt basiert auf den wichtigsten Erst- und Zweitlinienmedikamenten, die in den WHO-Leitlinien zur Tuberkulosebehandlung beschrieben sind, Makroliden und Aminoglykosiden, die häufig in den NTM-Behandlungsleitlinien verwendet werden, und die Resistenzstellen decken eine ganze Gruppe von Resistenzstellen im WHO-Mutationskatalog des Mycobacterium-tuberculosis-Komplexes sowie andere berichtete Resistenzgene und Mutationsstellen gemäß der Untersuchung und Statistik von hoch bewerteten Literaturen im In- und Ausland ab.
Die Typisierung basiert auf den in den NTM-Leitlinien des „Chinese Journal of Tuberculosis and Respiratory Diseases“ zusammengefassten NTM-Stämmen sowie auf dem Konsens von Experten. Die entwickelten Typisierungsprimer ermöglichen die Amplifikation, Sequenzierung und Annotation von über 190 NTM-Spezies.
Mithilfe gezielter Multiplex-PCR-Amplifikationstechnologie wurden die Genotypisierungs- und Arzneimittelresistenzgene von Mycobacterium amplifiziert und die Amplikonkombination der zu detektierenden Zielgene erhalten. Die amplifizierten Produkte können in Hochdurchsatz-Sequenzierungsbibliotheken der zweiten oder dritten Generation integriert werden. Alle Sequenzierungsplattformen der zweiten und dritten Generation ermöglichen eine Hochdurchsatzsequenzierung zur Gewinnung der Sequenzinformationen der Zielgene. Durch Vergleich mit bekannten Mutationen in der integrierten Referenzdatenbank (einschließlich des WHO-Mutationskatalogs des Mycobacterium-tuberculosis-Komplexes und dessen Zusammenhang mit Arzneimittelresistenz) wurden die mit Arzneimittelresistenz oder -empfindlichkeit gegenüber Tuberkulosemedikamenten assoziierten Mutationen identifiziert. In Kombination mit der selbstöffnenden Sputumprobenbehandlungslösung von Macro & Micro-Test wurde das Problem der geringen Nukleinsäureamplifikationseffizienz klinischer Sputumproben (zehnmal höher als bei herkömmlichen Methoden) gelöst, sodass die Sequenzierungserkennung von Arzneimittelresistenzen direkt auf klinische Sputumproben angewendet werden kann.
Produkterkennungsbereich
34Arzneimittelresistenz-bezogene Gene von18Antituberkulosemedikamente und6Es wurden NTM-Medikamente nachgewiesen, die Folgendes umfassten:297Resistenzstellen gegen Medikamente; zehn Arten von Mycobacterium tuberculosis und mehr190Es wurden verschiedene Arten von NTM nachgewiesen.
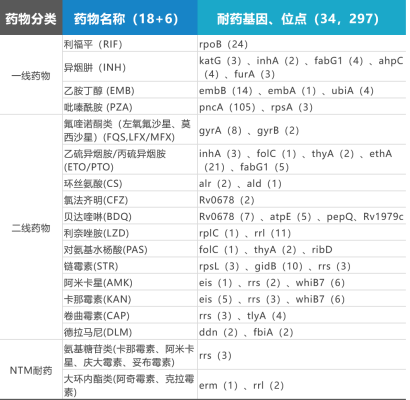
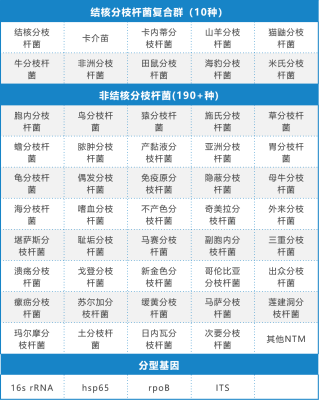
Tabelle 1: Informationen zu 18+6 Arzneimitteln +190+NTM
Produktvorteil
Hohe klinische Anpassungsfähigkeit: Die Sputumproben können ohne Kultur direkt mit einem Selbstverflüssigungsmittel untersucht werden.
Das experimentelle Vorgehen ist einfach: Die erste Amplifikationsrunde ist unkompliziert, und der Aufbau der Bibliothek ist in 3 Stunden abgeschlossen, was die Arbeitseffizienz steigert.
Umfassende Typisierung und Resistenzanalyse: Abdeckung der Typisierungs- und Resistenzstellen von MTB und NTM, die die wichtigsten klinischen Aspekte darstellen, genaue Typisierungs- und Resistenzerkennung, Unterstützung unabhängiger Analysesoftware und Generierung von Analyseberichten mit einem Klick.
Kompatibilität: Produktkompatibilität, Anpassung an gängige ILM- und MGI/ONT-Plattformen.
Produktspezifikation
| Produktcode | Produktname | Erkennungsplattform | Spezifikationen |
| HWTS-NGS-T001 | Kit zur Mykobakterien-Typisierung und zum Nachweis von Arzneimittelresistenzen (Multiplex-Amplifikationsverfahren) | ONT, Illumina, MGI, Salus pro | 16/96rxn |
Veröffentlichungsdatum: 23. Januar 2024
